DESTAQUE
Pesquisas premiadas com Nobel impulsionam avanços da Química no Brasil
A pesquisa dos laureados pelo Prêmio Nobel de Química de 2024 representa uma revolução na Química Medicinal e no desenvolvimento de fármacos. O prêmio dividido entre David Baker, da Universidade de Washington (EUA), e Demis Hassabis e John Jumper, do Google DeepMind (braço de inteligência artificial do Google), está relacionado ao desenvolvimento de um modelo de inteligência artificial (IA) e ferramenta computacional para prever e desenhar novas estruturas tridimensionais de proteínas.
As proteínas são responsáveis pelas funções bioquímicas no corpo humano. A contração ou a distensão muscular, a recepção de luz no olho, o transporte de oxigênio são alguns exemplos de funções que são realizadas por uma proteína. "A molécula em si é um fio de aminoácidos ligados em sequência, e o que determina a função que a proteína é capaz de fazer é sua estrutura tridimensional", explica o químico Fábio Gozzo, docente do Instituto de Química da Unicamp.
Determinar a sequência de aminoácidos não é uma tarefa difícil, mas determinar a estrutura tridimensional é bem mais complicado. Nas últimas três décadas, grupos de pesquisa vêm tentando encontrar um método computacional que consiga, a partir de uma dada sequência de aminoácidos, prever a estrutura tridimensional da proteína.
Em 1994, pesquisadores da Universidade de Maryland (EUA) lançaram a competição bianual internacional CASP (Critical Assessment of Protein Structure Prediction), com o objetivo de ajudar a desenvolver os métodos de identificação da estrutura da proteína a partir da sequência primária. "Os organizadores trabalhavam com proteínas cuja estrutura haviam determinado experimentalmente, passavam aos competidores (grupos que trabalhavam com modelagem) apenas a sequência de aminoácidos e depois comparavam os resultados dos modelos previstos", conta Gozzo.
|
|
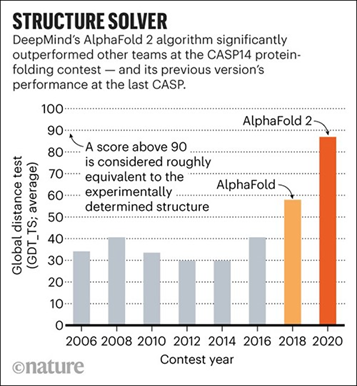
Gráfico mostra a evolução do melhor grupo de modelagem ao longo dos anos no concurso CASP. Até 2016, o grupo do David Baker acertava entre 30% e 40%. O Alphafold-2 acertou 60% na primeira versão e 90% na segunda versão
|
O grupo de David Baker era regularmente um dos que apresentava maior taxa de acerto (com seu software Rosetta, que chegava a 40% de acerto, resultado considerado muito bom) até que em 2018 a DeepMind -- então recentemente adquirida pelo Google – participou pela primeira vez, e ganhou com aproximadamente 60% de acerto. "No concurso seguinte, já com a versão Alphafold-2, o grupo de Hassabis e Jumper acertou 90% das estruturas, o que foi um assombro para a comunidade científica", relata o docente da Unicamp.
Enquanto a dupla do DeepMind foi laureada pelo método de IA de predição das estruturas das proteínas, Baker foi premiado por percorrer o caminho inverso. Em vez de achar a estrutura tridimensional a partir de uma sequência dada, ele imaginou qual a sequência de aminoácidos que forma uma proteína que precisar ter o formato capaz de bloquear uma enzima responsável por uma doença qualquer.
"Baker era um dos líderes na parte de predição até que surgisse o DeepMind, e ele colocou vários fundamentos nesse campo. Na parte inversa, ele sempre foi o líder, e já se especulava que ele ganharia o Nobel mais cedo ou mais tarde", observa Gozzo. "Ele teve um sucesso enorme, desenvolveu dezenas de proteínas funcionais, por exemplo para inibir o vírus da influenza, e abriu uma avenida para a descoberta de novos fármacos."
Em 2019, Gozzo tirou uma licença prêmio e foi trabalhar no Institute for Protein Design (IPT), criado por Baker, em Seattle. "No meu trabalho, eu uso espectrometria de massas para medir a distância tridimensional entre os aminoácidos na estrutura. Então entrei em contato com eles e fui passar uma temporada trabalhando com um professor associado do grupo, usando dados experimentais parciais para guiar a modelagem", conta.

|
|
Fabio Gozzo (Unicamp): "Baker era um dos líderes na parte de predição até que surgisse o DeepMind, e ele colocou vários fundamentos nesse campo. Já se especulava que ele ganharia o Nobel mais cedo ou mais tarde"
|
Ele descreve Baker como uma pessoa de muita energia. "Ele montou esse instituto, tem mais ou menos 150 pessoas trabalhando lá, e conversa com todos na bancada, sabe o que cada um está fazendo, discute problemas de computação, química, física, biologia, medicina, e sempre teve uma visão clara de que projetar proteínas para funções específicas era o futuro."

|
|
Carolina Horta Andrade (UFG): "A predição da estrutura tridimensional das proteínas com 95% de precisão é um avanço tremendo, que veio para revolucionar não somente a química medicinal, mas também a ambiental e todos os processos ligados à vida"
|
No Laboratório de Planejamento de Fármacos e Modelagem Molecular, da Faculdade de Farmácia da Universidade Federal de Goiás, a professora Carolina Horta Andrade, vem utilizando o Alphafold-2 desde 2020 em seu trabalho de desenvolvimento de candidatos a fármacos para doenças tropicais negligenciadas e viroses emergentes. "Os métodos experimentais são caros e demorados, então a predição da estrutura tridimensional das proteínas com 95% de precisão é um avanço tremendo, que veio para revolucionar não somente a química medicinal, mas também a ambiental e todos os processos ligados à vida", declara.
Com a inteligência artificial do Alphafold-2, o grupo da professora Carolina conseguiu modelar a estrutura tridimensional de uma proteína do vírus Chikungunya e do vírus Zika e, em colaboração com o grupo do Prof. Glaucius Oliva (IQSC-USP), obteve a estrutura tridimensional de uma das proteínas do vírus Zika e encontraram inibidores para essas proteínas.
"Este projeto ainda está aberto. Com a estrutura tridimensional conseguimos desenhar um novo composto e testamos na proteína do vírus. Conseguimos a atividade antiviral para ambos os vírus e estamos agora na etapa de otimização, para que obtenhamos compostos mais eficazes e seguros, mas também com propriedades farmacocinéticas otimizadas, que possam seguir para estudos pré-clínicos em modelos animais", descreve a docente.
Saiba mais:
Artigos do Prof. Gozzo:
"Characterizing protein conformers by cross-linking mass spectrometry and pattern recognition", Bioinformatics. 2021 Sep 29;37(18):3035-3037
"Structural complementarity of distance constraints obtained from chemical cross-linking and amino acid coevolution", Proteins. 2020 Apr;88(4):625-632
Artigos da Profa. Carolina:
https://www.nature.com/articles/s41598-022-14790-x
https://pubs.acs.org/doi/10.1021/acs.jcim.2c00596
CASP: https://predictioncenter.org/
Sobre o Alphafold: https://deepmind.google/discover/blog/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology/
Texto: Mario Henrique Viana (Assessoria de Imprensa da SBQ)
|